『nnU-Netの究極ガイド』
Ultimate Guide to nnU-Net
State of the Art nnU-Netの理解と独自のデータセットへの適用方法について知るために必要なすべてのこと。

私のケンブリッジ大学でのディープラーニングと神経科学の研究インターンシップでは、セマンティック画像セグメンテーションにおいて非常に強力な基準であるnnU-Netをよく使用しました。
しかし、このモデルとそのトレーニング方法を完全に理解するのに苦労し、インターネットであまり助けが見つからなかったです。今ではそれに慣れているので、このチュートリアルを作成しました。このチュートリアルは、このモデルの背後にあるものをよりよく理解するため、または独自のデータセットでの使用方法を知るためにお手伝いできることを願っています。
このガイドの中で、以下のことを学びます:
- nnU-Netの主要な貢献の簡潔な概要を作成する。
- nnU-Netを独自のデータセットに適用する方法を学ぶ。
すべてのコードはこのGoogle Collabノートブックで利用できます。
- Google DeepMindの研究者たちは、RT-2という新しいビジョン・言語・行動(VLA)モデルを紹介しましたこのモデルは、ウェブデータとロボットデータの両方から学習し、それを行動に変えます
- 「データフレームのマージに使用される3つのPandas関数」
- 『AWS SageMaker Data Wranglerの新機能でデータ準備を最適化する』
この作業には多大な時間と努力がかかりました。このコンテンツが価値あるものと考えられる場合は、私をフォローしてその可視性を高め、より多くのチュートリアルの作成に支援していただけると幸いです!
nnU-Netの概略
Image Segmentationにおける最先端のモデルとして認識されているnnU-Netは、2Dおよび3D画像処理の両方において強力な性能を発揮します。その性能は非常に堅牢であり、新しいコンピュータビジョンアーキテクチャのベンチマークとして強力な基準となります。要するに、新しいコンピュータビジョンモデルの開発の世界に足を踏み入れる場合、nnU-Netを目標とするべき存在として考えてください。
この強力なツールは、2015年にデビューしたU-Netモデルに基づいています(U-Netのチュートリアルの一つはこちら:はじめてのU-Net)。”nnU-Net”という名称は、その設計が革新的なアーキテクチャの変更を導入しないことを示しています。代わりに、既存のU-Net構造を取り入れ、一連の巧妙な最適化戦略を使用してその全ポテンシャルを引き出します。
近年の多くのニューラルネットワークとは異なり、nnU-Netは残差接続、密な接続、または注意メカニズムに依存しません。その強みは、リサンプリング、正規化、損失関数の適切な選択、オプティマイザの設定、データ拡張、パッチベースの推論、モデル間のアンサンブルなどの技術を含む緻密な最適化戦略にあります。この包括的なアプローチにより、nnU-Netは元のU-Netアーキテクチャで実現可能な領域を広げることができます。
nnU-Net内の多様なアーキテクチャの探索
一つのエンティティのように見えるかもしれませんが、nnU-Netは実際には3つの異なるタイプのU-Netを指す総称です:
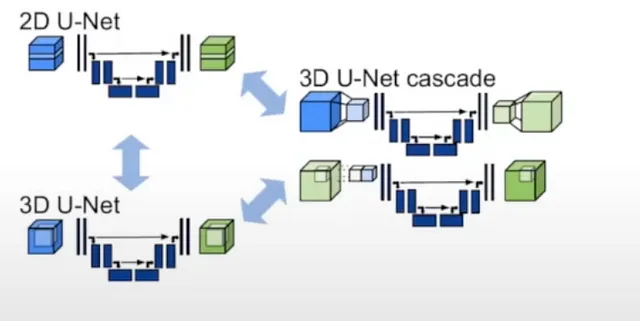
- 2D U-Net:おそらく最もよく知られたバリアントで、これは直接2D画像上で動作します。
- 3D U-Net:これは2D U-Netの拡張であり、3D畳み込みを適用することで直接3D画像を処理することができます。
- U-Net Cascade:このモデルは低解像度のセグメンテーションを生成し、その後それを洗練させます。
これらのアーキテクチャはそれぞれ独自の強みを持ち、避けることができない制限もあります。
例えば、3D画像セグメンテーションのために2D U-Netを使用することは直感に反するように思えますが、実際には非常に効果的です。これは、3Dボリュームを2Dの平面にスライスすることによって実現されます。
3D U-Netはより洗練されたように思えるかもしれませんが、パラメータの数が多いため、常に最も効率的な解決策ではありません。特に、3D U-Netは異方性に苦労することがよくあります。異方性は、異なる軸に沿って空間解像度が異なる場合に発生します(たとえば、x軸方向に1mm、z軸方向に1.2mmの場合など)。
U-Netのカスケードバリアントは、大きな画像サイズを扱うときに特に便利です。まず、画像を凝縮するための初期モデルを使用し、それに続いて低解像度のセグメンテーションを出力する標準の3D U-Netを使用します。生成された予測値は拡大され、洗練された包括的な出力が得られます。
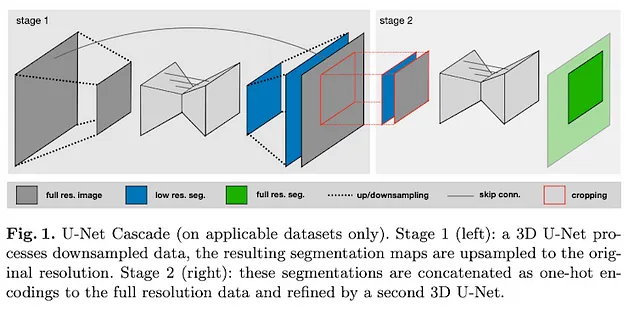
通常、この手法では、nnU-Netフレームワーク内で3つのモデルバリアントをすべてトレーニングします。次のステップは、3つのうち最も優れたパフォーマンスを持つモデルを選択するか、アンサンブル技術を使用することです。このような技術の1つは、2Dと3D U-Netの両方の予測値を統合することです。
ただし、この手順はかなり時間がかかること(そしてGPUクレジットが必要なためお金もかかること)に注意する価値があります。制約が1つのモデルのトレーニングのみを許す場合は、心配しないでください。アンサンブルモデルは非常にわずかな利益しかもたらしませんので、1つのモデルのみをトレーニングすることを選択できます。
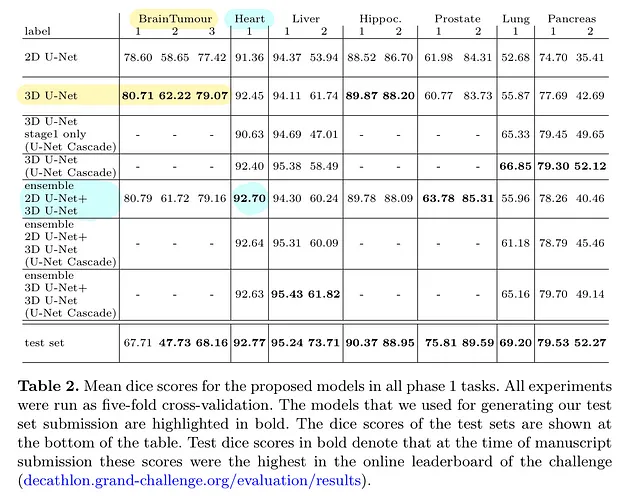
この表は、特定のデータセットに関連する最も優れたモデルバリアントを示しています:
ネットワークトポロジの動的適応
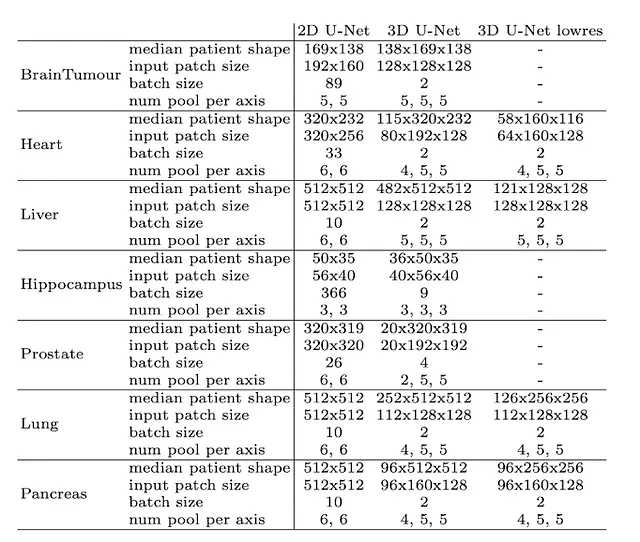
画像サイズの大きな違い(肝臓画像の中央形状が482×512×512であるのに対し、海馬画像の中央形状が36×50×35であることを考慮してください)、nnU-Netは入力パッチのサイズと各軸ごとのプーリング操作の数を知的に適応させます。これは、データセットごとに畳み込み層の数を自動的に調整することを意味し、空間情報の効果的な集約を容易にします。さらに、画像の幾何学的な違いに適応するだけでなく、利用可能なメモリなどの技術的な制約も考慮に入れます。
重要なポイントは、モデルが全体の画像ではなく、重なり合う領域を持つ慎重に抽出されたパッチ上でセグメンテーションを行うことです。これらのパッチ上の予測値は後で平均化され、最終的なセグメンテーション出力が生成されます。
ただし、大きなパッチを持つことはメモリ使用量が増え、バッチサイズもメモリを消費します。トレードオフは、バッチサイズ(最適化にのみ有用)ではなく、パッチサイズ(モデルの容量)を常に優先するということです。
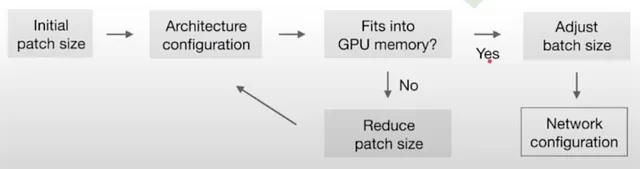
以下は、最適なパッチサイズとバッチサイズを計算するために使用されるヒューリスティックアルゴリズムです:
そして、これが異なるデータセットと入力次元の場合の外観です:
素晴らしい!では、nnU-Netで使用されるすべての技術について簡単に説明しましょう:
トレーニング
すべてのモデルはゼロからトレーニングされ、トレーニングセットで5つのフォールド交差検証を使用して評価されます。つまり、元のトレーニングデータセットがランダムに5つの等しい部分、または「フォールド」に分割されます。この交差検証プロセスでは、これらのうち4つのフォールドがモデルのトレーニングに使用され、残りの1つのフォールドが評価またはテストに使用されます。このプロセスは5回繰り返され、5つのフォールドのそれぞれが正確に1回だけ評価セットとして使用されます。
損失関数には、Dice損失とクロスエントロピー損失の組み合わせを使用しています。これは画像セグメンテーションで非常に頻繁に使用される損失関数です。Dice損失の詳細については、V-NetのDice損失、U-Netの兄弟に関する詳細を参照してください。
データ拡張技術
nnU-Netは非常に強力なデータ拡張パイプラインを持っています。著者はランダムな回転、ランダムなスケーリング、ランダムな弾性変形、ガンマ補正、ミラーリングを使用しています。
NB:ソースコードを変更することで独自の変換を追加できます

パッチベースの推論
したがって、モデルは直接フル解像度の画像に予測を行わず、抽出されたパッチに対して予測を行い、それを集計します。
それは次のようになります:
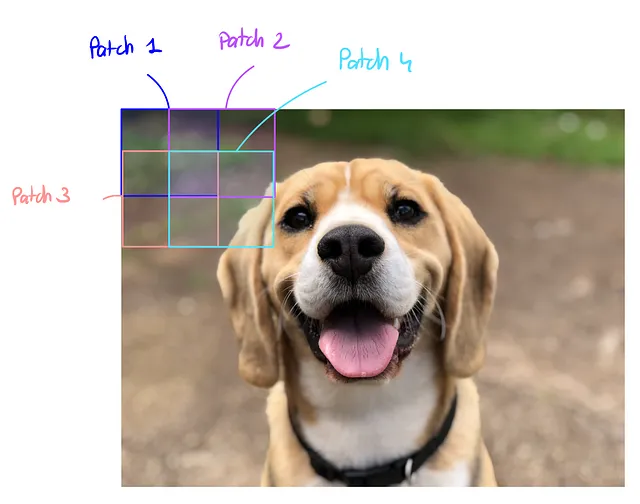
NB:画像の中央のパッチは、より多くの情報を含んでおり、モデルのパフォーマンスが向上するため、より重みが与えられます
ペアワイズモデルアンサンブリング
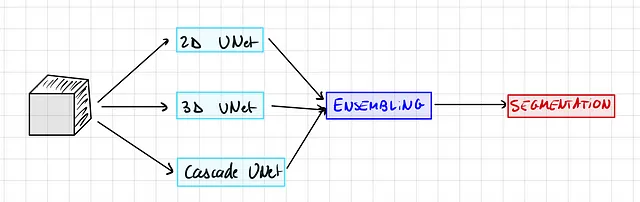
さて、よく覚えているかと思いますが、2D、3D、およびカスケードの3つの異なるモデルをトレーニングできますが、推論を行う際には一度に1つのモデルしか使用できないですよね?
しかし、異なるモデルにはそれぞれ異なる強みと弱みがあります。したがって、実際には複数のモデルの予測を組み合わせることができ、もし1つのモデルが非常に自信を持っている場合は、その予測を優先することができます。
nnU-Netは、利用可能な3つのモデルの中から2つのモデルのすべての組み合わせをテストし、最適な組み合わせを選択します。
実際には、2つの方法があります:
ハード投票:各ピクセルごとに、2つのモデルが出力した確率を調べ、最も高い確率のクラスを取ります。
ソフト投票:各ピクセルごとに、モデルの確率を平均化し、最大の確率のクラスを取ります。
実装の実践
始める前に、ここでデータセットをダウンロードし、Google Collabのノートブックに従ってください。
最初のパートについて何も理解できなかった場合は心配しないでください、これは実践的なパートです。私に従ってください、そして最高の結果を得ることができます。
モデルをトレーニングするためにはGPUが必要です。ローカルで行うか、Google Collabで行うことができます。ランタイムを変更してGPUに設定するのを忘れないでください
まず、入力画像とそれに対応するセグメンテーションを持つデータセットが必要です。3D脳セグメンテーションのための準備済みのデータセットをダウンロードして、自分のデータセットで置き換えることができます。
データのダウンロード
まず、データをダウンロードして、セグメンテーションを含む「input」と「ground_truth」という2つのフォルダに配置する必要があります。
チュートリアルの残りの部分では、画像セグメンテーションのためのMindBoggleデータセットを使用します。これをGoogleドライブからダウンロードできます:
私たちは脳の3D MRIスキャンを与えられ、白質と灰白質をセグメンテーションしたいと考えています:
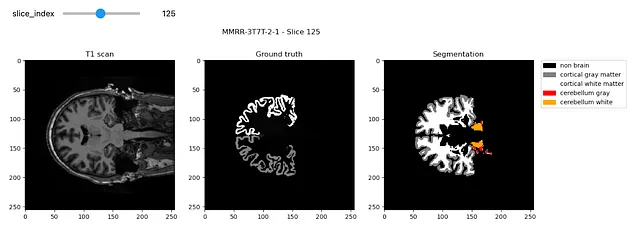
このように見えるはずです:
メインディレクトリの設定
Google Colabで実行する場合は、collab = Trueとし、それ以外の場合はcollab = Falseとします
collab = Trueimport osimport shutil#librariesfrom collections import OrderedDictimport jsonimport numpy as np#データセットの可視化import matplotlib.pyplot as pltimport nibabel as nibif collab: from google.colab import drive drive.flush_and_unmount() drive.mount('/content/drive', force_remount=True) # "neurosciences-segmentation"をプロジェクトフォルダーの名前に変更してください root_dir = "/content/drive/MyDrive/neurosciences-segmentation"else: # 親ディレクトリのディレクトリを取得 root_dir = os.getcwd()input_dir = os.path.join(root_dir, 'data/input')segmentation_dir = os.path.join(root_dir, 'data/ground_truth')my_nnunet_dir = os.path.join(root_dir,'my_nnunet')print(my_nnunet_dir)ここで、フォルダを作成する関数を定義します:
def make_if_dont_exist(folder_path,overwrite=False): """ フォルダが存在しない場合は作成します 入力: folder_path: 作成するフォルダの相対パス over_write: (デフォルト: False) Trueの場合、既存のフォルダを上書きします """ if os.path.exists(folder_path): if not overwrite: print(f'{folder_path} は既に存在します。') else: print(f"{folder_path} を上書きします。") shutil.rmtree(folder_path) os.makedirs(folder_path) else: os.makedirs(folder_path) print(f"{folder_path} を作成しました!")そして、この関数を使用して、すべてが保存される「my_nnunet」フォルダを作成します
os.chdir(root_dir)make_if_dont_exist('my_nnunet', overwrite=False)os.chdir('my_nnunet')print(f"現在の作業ディレクトリ: {os.getcwd()}")ライブラリのインストール
ここで、すべての要件をインストールします。まず、nnunetライブラリをインストールします。ノートブックの場合はセルで実行します:
!pip install nnunetそれ以外の場合は、ターミナルから直接nnunetをインストールできます:
pip install nnunet次に、nnUnet gitリポジトリとNVIDIA apexをクローンします。これにはトレーニングスクリプトとGPUアクセラレータが含まれています。
!git clone https://github.com/MIC-DKFZ/nnUNet.git!git clone https://github.com/NVIDIA/apex# リポジトリディレクトリはgithubフォルダのパスですrespository_dir = os.path.join(my_nnunet_dir,'nnUNet')os.chdir(respository_dir)!pip install -e!pip install --upgrade git+https://github.com/nanohanno/hiddenlayer.git@bugfix/get_trace_graph#egg=hiddenlayerフォルダの作成
nnUnetには、フォルダの非常に特定の構造が必要です。
task_name = 'Task001' #異なるタスク名に変更する場合はここを変更します# 必要なすべてのパスを定義しますnnunet_dir = "nnUNet/nnunet/nnUNet_raw_data_base/nnUNet_raw_data"task_folder_name = os.path.join(nnunet_dir,task_name) train_image_dir = os.path.join(task_folder_name,'imagesTr') # トレーニング画像へのパストrain_label_dir = os.path.join(task_folder_name,'labelsTr') # トレーニングラベルへのパストest_dir = os.path.join(task_folder_name,'imagesTs') # テスト画像へのパスmain_dir = os.path.join(my_nnunet_dir,'nnUNet/nnunet') # メインディレクトリへのパストrained_model_dir = os.path.join(main_dir, 'nnUNet_trained_models') # トレーニング済みモデルへのパス元々、nnU-Netは異なるタスクを持つデカスロンチャレンジ向けに設計されました。異なるタスクがある場合は、すべてのタスクに対してこのセルを実行してください。
# すべてのフォルダを作成しますoverwrite = False # フォルダを上書きする場合は True に設定してくださいmake_if_dont_exist(task_folder_name,overwrite = overwrite)make_if_dont_exist(train_image_dir, overwrite = overwrite)make_if_dont_exist(train_label_dir, overwrite = overwrite)make_if_dont_exist(test_dir,overwrite= overwrite)make_if_dont_exist(trained_model_dir, overwrite=overwrite)これで次のような構造になるはずです:
環境変数の設定
スクリプトは、生データの場所、前処理済みデータの場所、および結果を保存する場所を知る必要があります。
os.environ['nnUNet_raw_data_base'] = os.path.join(main_dir,'nnUNet_raw_data_base')os.environ['nnUNet_preprocessed'] = os.path.join(main_dir,'preprocessed')os.environ['RESULTS_FOLDER'] = trained_model_dir適切なリポジトリにファイルを移動する
nnunetフォルダの適切なリポジトリに画像を移動する関数を定義します:
def copy_and_rename(old_location,old_file_name,new_location,new_filename,delete_original = False): shutil.copy(os.path.join(old_location,old_file_name),new_location) os.rename(os.path.join(new_location,old_file_name),os.path.join(new_location,new_filename)) if delete_original: os.remove(os.path.join(old_location,old_file_name))これで、入力画像と正解画像に対してこの関数を実行しましょう:
list_of_all_files = os.listdir(segmentation_dir)list_of_all_files = [file_name for file_name in list_of_all_files if file_name.endswith('.nii.gz')]for file_name in list_of_all_files: copy_and_rename(input_dir,file_name,train_image_dir,file_name) copy_and_rename(segmentation_dir,file_name,train_label_dir,file_name)これで、ファイルの名前をnnUnet形式で受け入れられるように変更する必要があります。例えば、subject.nii.gzはsubject_0000.nii.gzになります。
def check_modality(filename): """ モダリティの存在をチェックします。モダリティが見つからない場合はFalse、それ以外の場合はTrueを返します。 """ end = filename.find('.nii.gz') modality = filename[end-4:end] for mod in modality: if not(ord(mod)>=48 and ord(mod)<=57): # 0から9の数字でない場合 return False return Truedef rename_for_single_modality(directory): for file in os.listdir(directory): if check_modality(file)==False: new_name = file[:file.find('.nii.gz')]+"_0000.nii.gz" os.rename(os.path.join(directory,file),os.path.join(directory,new_name)) print(f"Renamed to {new_name}") else: print(f"Modality present: {file}")rename_for_single_modality(train_image_dir)# rename_for_single_modality(test_dir)JSONファイルの設定
ほとんど完了です!
主に2つの項目を変更する必要があります:
- モダリティ(CTまたはMRIの場合はこれが正規化を変更します)
- ラベル: 自分自身のクラスを入力してください
overwrite_json_file = True # dataset.jsonファイルを上書きする場合はTrueに設定してくださいjson_file_exist = Falseif os.path.exists(os.path.join(task_folder_name,'dataset.json')): print('dataset.jsonはすでに存在しています!') json_file_exist = Trueif json_file_exist==False or overwrite_json_file: json_dict = OrderedDict() json_dict['name'] = task_name json_dict['description'] = "Segmentation of T1 Scans from MindBoggle" json_dict['tensorImageSize'] = "3D" json_dict['reference'] = "see challenge website" json_dict['licence'] = "see challenge website" json_dict['release'] = "0.0" ######################## ここを変更してください ######################## # 複数のモダリティを指定できます json_dict['modality'] = { "0": "MRI" } # データセット内のすべてのラベルについてlabels+1を指定する必要があります json_dict['labels'] = { "0": "Non Brain", "1": "Cortical gray matter", "2": "Cortical White matter", "3" : "Cerebellum gray ", "4" : "Cerebellum white" } ############################################################# train_ids = os.listdir(train_label_dir) test_ids = os.listdir(test_dir) json_dict['numTraining'] = len(train_ids) json_dict['numTest'] = len(test_ids) # データセット.jsonにはトレーニング画像とラベルにモダリティは含まれません json_dict['training'] = [{'image': "./imagesTr/%s" % i, "label": "./labelsTr/%s" % i} for i in train_ids] # テスト画像の名前からモダリティを削除してデータセット.jsonに保存します json_dict['test'] = ["./imagesTs/%s" % (i[:i.find("_0000")]+'.nii.gz') for i in test_ids] with open(os.path.join(task_folder_name,"dataset.json"), 'w') as f: json.dump(json_dict, f, indent=4, sort_keys=True) if os.path.exists(os.path.join(task_folder_name,'dataset.json')): if json_file_exist==False: print('dataset.jsonが作成されました!') else: print('dataset.jsonが上書きされました!')nnU-Net形式にデータを前処理する
これにより、nnU-Net形式のデータセットが作成されます
# -t 1は「Task001」を意味し、異なるタスクがある場合は変更してください!nnUNet_plan_and_preprocess -t 1 --verify_dataset_integrityモデルを訓練する
モデルの訓練には準備ができました!
3D U-Netを訓練するには:
# 3Dフル解像度U-Netを訓練する!nnUNet_train 3d_fullres nnUNetTrainerV2 1 0 --npz 2D U-Netを訓練するには:
# 2D U-Netを訓練する!nnUNet_train 2d nnUNetTrainerV2 1 0 --npzカスケードモデルを訓練するには:
# 3D U-Netカスケードを訓練する!nnUNet_train 3d_lowres nnUNetTrainerV2CascadeFullRes 1 0 --npz!nnUNet_train 3d_fullres nnUNetTrainerV2CascadeFullRes 1 0 --npz注意:訓練を一時停止して再開したい場合は、「-c」を末尾に追加してください。
例えば:
# 3Dフル解像度U-Netを訓練する!nnUNet_train 3d_fullres nnUNetTrainerV2 1 0 --npz 推論
今、推論を実行できます:
result_dir = os.path.join(task_folder_name, 'nnUNet_Prediction_Results')make_if_dont_exist(result_dir, overwrite=True)# -iは入力フォルダです# -oは予測結果を保存する場所です# -t 1はタスク1を意味し、異なるタスク番号の場合は変更してください# -m 2d、または -m 3d_fullres、または -m 3d_cascade_fullresを使用してください!nnUNet_predict -i /content/drive/MyDrive/neurosciences-segmentation/my_nnunet/nnUNet/nnunet/nnUNet_raw_data_base/nnUNet_raw_data/Task001/imagesTs -o /content/drive/MyDrive/neurosciences-segmentation/my_nnunet/nnUNet/nnunet/nnUNet_raw_data_base/nnUNet_raw_data/Task001/nnUNet_Prediction_Results -t 1 -tr nnUNetTrainerV2 -m 2d -f 0 --num_threads_preprocessing 1予測の可視化
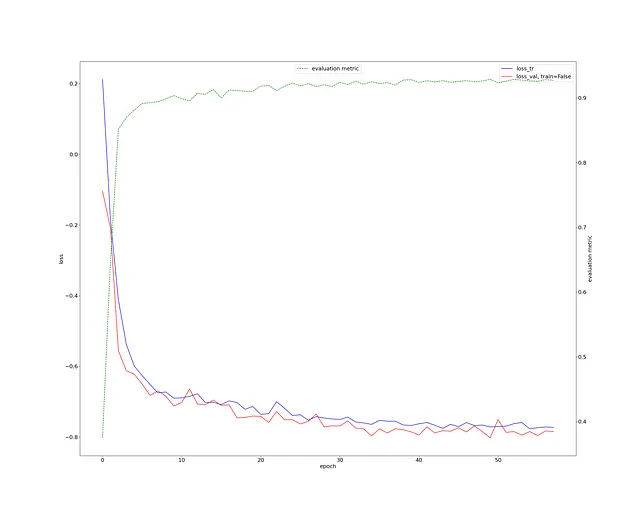
まず、訓練損失を確認しましょう。非常に健全であり、Diceスコアは0.9以上です(緑の曲線)。
これは非常に少ない作業と3D脳画像セグメンテーションタスクに対して非常に優れています。
サンプルを1つ見てみましょう:
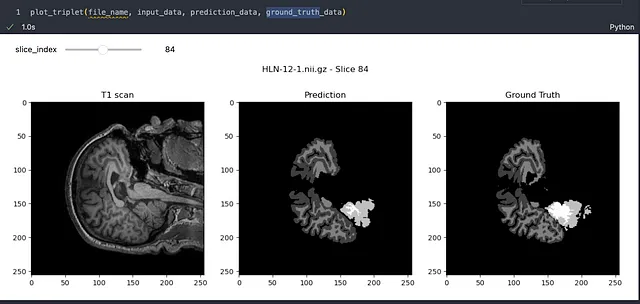
結果は本当に素晴らしいです!モデルは脳画像のセグメンテーションを高い精度で効果的に学習していることが明確です。細かな欠点があるかもしれませんが、画像セグメンテーションの分野は急速に進歩しており、完璧に向かって大きな進歩を遂げています。
将来的には、nnU-Netのパフォーマンスをさらに最適化する余地がありますが、それは別の記事の話題となります。
この記事が有益で興味深いと感じた場合は、深層学習の世界におけるより詳細な探求についての情報を提供するために、フォローをご検討ください。皆さんのサポートは、共同理解を助けるコンテンツの制作を続けるために役立ちます。
フィードバックがある場合、共有したいアイデアがある場合、一緒に働きたい場合、または単にこんにちはと言いたい場合は、以下のフォームに記入して、会話を始めましょう。
こんにちはを言ってください 🌿
もっと見るために、拍手を残したりフォローしたりすることを躊躇しないでください!
参考文献
- Ronneberger, O., Fischer, P., & Brox, T. (2015). U-net: Convolutional networks for biomedical image segmentation. In International Conference on Medical image computing and computer-assisted intervention (pp. 234–241). Springer, Cham.
- Isensee, F., Jaeger, P. F., Kohl, S. A., Petersen, J., & Maier-Hein, K. H. (2021). nnU-Net: a self-configuring method for deep learning-based biomedical image segmentation. Nature Methods, 18(2), 203–211.
- Ioffe, S., & Szegedy, C. (2015). Batch normalization: Accelerating deep network training by reducing internal covariate shift. arXiv preprint arXiv:1502.03167.
- Ulyanov, D., Vedaldi, A., & Lempitsky, V. (2016). Instance normalization: The missing ingredient for fast stylization. arXiv preprint arXiv:1607.08022.
- MindBoggleデータセット
We will continue to update VoAGI; if you have any questions or suggestions, please contact us!
Was this article helpful?
93 out of 132 found this helpful
Related articles
