「Pythonによる(バイオ)イメージ解析:Matplotlibを使用して顕微鏡画像を読み込み、ロードする」
Pythonでの(バイオ)イメージ解析:Matplotlibで顕微鏡画像を読み込み、ロードする
過去20年間、光顕微鏡の分野は画期的な技術の導入により、共焦点レーザースキャン顕微鏡(CLSM)、多光子レーザースキャン顕微鏡(MPLSM)、光シート蛍光顕微鏡(LSFM)、および超解像顕微鏡などの画期的な技術の導入により、著しい進歩を遂げてきました[1]。これらの技術は、深さの透過性、生存性、および解像度が向上した画像を取得する能力を大幅に向上させました。しかし、これらの画期的な進展に伴い、生成される膨大な量のデータを管理するという課題も生じました。これらの複雑なデータセットを効果的に視覚化し、分析し、普及させることは、これらの最先端顕微鏡技術のフルポテンシャルを引き出す上で重要な要素となっています。このチュートリアルでは、強力なMatplotlib Pythonライブラリを使用して、複雑な2D、3D、4D、および5D光顕微鏡画像を読み取り、視覚化する方法について探求します。
目次
- 1.1 画像とピクセル
- 1.2 チャンネル
- 1.3 4D画像(x、y、チャンネル、Zスライス)
- 1.4 投影
- 1.5 5D画像(x、y、チャンネル、Zスライス、時間)
- 1.6 結論
- 1.7 参考文献
# ライブラリのインポートimport numpy as npimport matplotlib.pyplot as pltimport skimage as skfrom matplotlib_scalebar.scalebar import ScaleBar画像とピクセル
顕微鏡画像はデジタルで記録され、そのために最終画像を形成する光子のフラックスは、幾何学的なサブユニットである画素(ピクセル)に分割されなければなりません[2]。コンピュータにとって、各ピクセルは単なる数値です(図1)。そして、画像データが表示されるとき、ピクセルの値は通常、正方形に変換されます。これらの正方形は、画像内容を素早く把握するのに役立つ視覚化手法にすぎません[3]。
![図1. 真菌*Aspergillus nidulans*のコニディオスポアにおけるPilA-GFPエイソソームタンパク質[5]、[6]の細胞内局在の顕微鏡画像。拡大された領域(赤いインセット)にはピクセルの値が表示されています。この画像はLeica TCS SP8 MP多光子顕微鏡を使用してキャプチャされました(著者提供の図)。](https://miro.medium.com/v2/format:webp/1*L4Luyzm04ji5moFMT36iEw.png)
顕微鏡および特に蛍光では、通常、画像はカラー情報なしで取得され(モノクロームカメラやフォトマルチプライヤを使用)、モノクロームの偽色LUTを使用して表示されます。ルックアップテーブル(LUT;場合によってはカラーマップとも呼ばれます)は、グレースケールの値がカラー値に変換される方法を決定します。顕微鏡画像を視覚化するために、私たちはしばしばグレーシェード(グレーカラーマップ)を使用します。また、読者の視覚感度を低い特徴に対して高めるために、画像を反転させ、暗い値を明るく、明るい値を暗くマッピングすることもあります[4]。
すべての公開された顕微鏡画像には、画像中の構造や特徴のサイズの視覚的な参照としてスケールバーが含まれている必要があります。
# scikit-imageを使用して画像を読み込むimg = sk.io.imread("Single_channel_eisosome.tif")# 画像の寸法を表示するprint(f"画像の寸法は:{img.shape[0]} x {img.shape[1]} ピクセル")# 2つのサブプロットを持つフィギュアを作成するfig, axs = plt.subplots(1, 2, figsize=(6, 4))# 最初のサブプロットにグレースケール画像をプロットするaxs[0].imshow(img,cmap="gray")axs[0].axis('off')# サブプロットにスケールバーを追加するscalebar = ScaleBar(0.0721501, 'um', length_fraction=0.2, height_fraction=0.02, location='lower right')axs[0].add_artist(scalebar)axs[0].set_title("グレースケール画像")# 2番目のサブプロットに反転したグレースケール画像をプロットするaxs[1].imshow(img,cmap="gray_r")axs[1].axis('off')# サブプロットにスケールバーを追加するscalebar = ScaleBar(0.0721501, 'um', length_fraction=0.2, height_fraction=0.02, location='lower right')axs[1].add_artist(scalebar)axs[1].set_title("反転画像")# サブプロット間のスペーシングを調整するplt.tight_layout()画像の寸法は:589 x 589 ピクセル
チャネル
顕微鏡法では、異なるチャネルはサンプル内の特定の成分や構造を捉え、可視化するために使用される異なるスペクトル範囲を表します。各チャネルは、特定の波長範囲や発光スペクトルに対応しており、サンプル内の異なる分子、構造、またはイベントを同時にイメージングすることができます。視覚的な認識を最適化するために、ほとんどの蛍光イメージングアプリケーションでは、別々の白黒チャネルの使用が推奨されています。最終的なマージされたカラーイメージを作成して、一般的な色覚障害を持つ読者にもアクセシビリティを提供することができます。以下のコードでは、A. nidulansのコニディオスポア内でPilA-GFP(グレースケールルックアップテーブルを使用)とヒストンH1-mRFP(グレースケールルックアップテーブルを使用)のサブセルラー局在を可視化し、オーバーレイを表示しています。オーバーレイでは、PilAはマゼンタで表示され、Histone H1は緑で表示されます[7]。
# scikit-imageを使用して画像を読み込む
img = sk.io.imread("3_channel_eisosome.tif")
# 画像の寸法を出力する
print(f"画像の寸法は{img.shape[0]} x {img.shape[1]}ピクセルで、{img.shape[2]}チャネルです")
# チャネルを分割する
h1_histone = img[:,:,0]
pila = img[:,:,1]
brightfield = img[:,:,2]
# 4つのサブプロットを持つ図を作成する
fig, axs = plt.subplots(1, 4, figsize=(12, 4))
# 最初のサブプロットにH1ヒストンをプロットする
axs[0].imshow(h1_histone,cmap="gray")
axs[0].axis('off')
axs[0].set_title("H1ヒストン(グレースケールイメージ)")
# 2番目のサブプロットにPilAをプロットする
axs[1].imshow(pila,cmap="gray")
axs[1].axis('off')
axs[1].set_title("PilAタンパク質(グレースケールイメージ)")
# 3番目のサブプロットに明視野画像をプロットする
axs[2].imshow(brightfield,cmap="gray")
axs[2].axis('off')
scalebar = ScaleBar(0.0721501, 'um', length_fraction=0.2, height_fraction=0.02, location='lower right')
axs[2].add_artist(scalebar)
axs[2].set_title("明視野")
# 2Dスライスを3D配列に変換する
h1_histone_3d = np.atleast_3d(img[:,:,0]) # 2Dスライスを3D配列に変換する
pila_3d = np.atleast_3d(img[:,:,1]) # 2Dスライスを3D配列に変換する
# 色を定義する
color_h1 = np.asarray([1, 0, 1]).reshape((1, 1, -1)) # 1x1x3の配列を作成する(マゼンタ色)
color_pila = np.asarray([0, 1, 0]).reshape((1, 1, -1)) # 1x1x3の配列を作成する(緑色)
# 画像内の各ピクセルに色を乗算する
h1_magenta = h1_histone_3d * color_h1
pila_green = pila_3d * color_pila
# 色のチャネルをマージする
merged = np.clip(h1_magenta + pila_green,0,255)
# 4番目のサブプロットにマージされたチャネルをプロットする
axs[3].imshow(merged)
axs[3].axis('off')
axs[3].set_title("マージされたチャネル")
# サブプロット間のスペーシングを調整する
plt.tight_layout()
画像の寸法は589 x 589ピクセルで、3チャネルです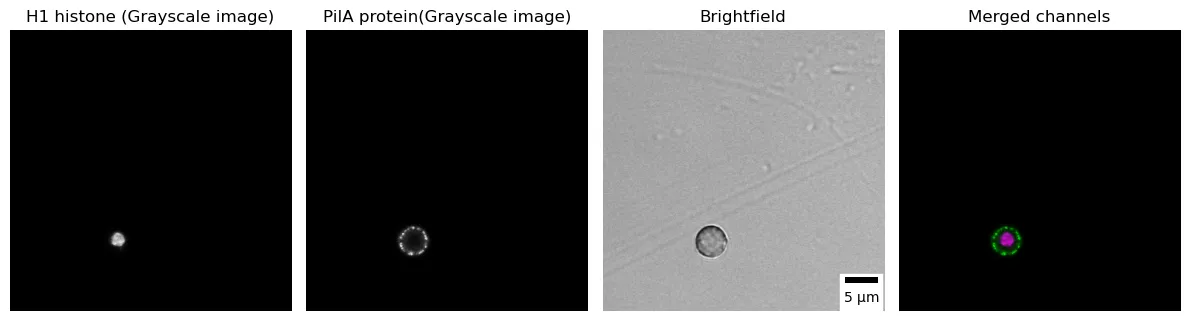
4Dイメージ(x、y、チャネル、およびZスライス)
共焦点顕微鏡法では、Z軸に沿った異なる焦点面での画像の「スライス」(Zスタック)を頻繁にキャプチャします。これらのスライスは、一緒に積み重ねることで3Dイメージに再構成されます。Zスタックは、サンプル内の構造の空間分布、形態、および関係に関する貴重な洞察を提供します。以下のコードでは、A. nidulansのコニディオスポア内でPilA-GFPとHistone H1-mRFPのサブセルラー局在を可視化するために、Z軸に沿って4つの「スライス」をキャプチャしています(実際には、以下の画像はハイパースタックを表しており、x、y、z、およびチャネルから構成される4Dの組成です)。このアプローチにより、これらのタンパク質の分布と関係を三次元で研究することができます。
# scikit-imageを使用して画像を読み込む
img = sk.io.imread("3_channel_zstack_eisosome.tif")
# 画像の次元を出力する
print(f"画像の次元は{img.shape[1]} x {img.shape[2]}ピクセルで、チャンネル数は{img.shape[3]}、スライス数は{img.shape[0]}です")
# チャンネルを分割する
h1_histone = img[:,:,:,0]
pila = img[:,:,:,1]
brightfield = img[:,:,:,2]
# サブプロットを持つ図を作成する
fig, axs = plt.subplots(3, 4, figsize=(12, 7))
# H1ヒストンチャンネルの各スライスをプロットする
for i in range(img.shape[0]):
axs[0][i].imshow(h1_histone[i,:,:],cmap="gray")
axs[0][i].axis('off')
axs[0][i].set_title(f"(Z = {i+1}) H1ヒストン")
scalebar = ScaleBar(0.0721501, 'um', length_fraction=0.2, height_fraction=0.02, location='lower right')
axs[0, -1].add_artist(scalebar)
# PilAタンパク質チャンネルの各スライスをプロットする
for i in range(img.shape[0]):
axs[1][i].imshow(pila[i,:,:],cmap="gray")
axs[1][i].axis('off')
axs[1][i].set_title(f"(Z = {i+1}) PilAタンパク質")
scalebar = ScaleBar(0.0721501, 'um', length_fraction=0.2, height_fraction=0.02, location='lower right')
axs[1, -1].add_artist(scalebar)
# H1ヒストンとPilAタンパク質チャンネルのマージ画像を作成する
for i in range(img.shape[0]):
h1_histone_3d = np.atleast_3d(h1_histone[i,:,:])
pila_3d = np.atleast_3d(pila[i,:,:])
color_h1 = np.asarray([1, 0, 1]).reshape((1, 1, -1))
color_pila = np.asarray([0, 1, 0]).reshape((1, 1, -1))
h1_magenta = h1_histone_3d * color_h1
pila_green = pila_3d * color_pila
merged = np.clip(h1_magenta + pila_green,0,255)
axs[2][i].imshow(merged)
axs[2][i].axis('off')
axs[2][i].set_title(rf"(Z = {i+1}) H1とPilA")
scalebar = ScaleBar(0.0721501, 'um', length_fraction=0.2, height_fraction=0.02, location='lower right')
axs[2, -1].add_artist(scalebar)
# サブプロット間のスペースを調整する
plt.tight_layout()
# 画像の次元を出力する
print("画像の次元は589 x 589ピクセルで、チャンネル数は3、スライス数は4です")

プロジェクション
Zスタックを視覚化する方法の1つは、上記のコードで示されているように、各スライスを個別に調べることです。ただし、複数のスライスを扱う場合、この方法は煩雑になります。Zスタックの情報を要約する効率的な方法は、zプロジェクションを計算することです [3]。zプロジェクションを実行することで、特定の質問に基づいてさまざまなタイプのプロジェクションを生成することができます。たとえば、最大プロジェクション、最小プロジェクション、平均プロジェクション、標準偏差プロジェクションなどを取得することができます。最大プロジェクションでは、最大の強度を持つ特徴をキャプチャするために、特定の軸(通常はz軸)に沿って2D画像が作成されます。同様に、平均、最小、および標準偏差プロジェクションでは、特定の軸に沿って強度の平均、最小、または拡散を表す2D画像が生成されます。
# scikit-imageを使用して画像を読み込む
img = sk.io.imread("3_channel_zstack_eisosome.tif")
# チャンネルを分割する
h1_histone = img[:,:,:,0]
pila = img[:,:,:,1]
brightfield = img[:,:,:,2]
# サブプロットを持つ図を作成する
fig, axs = plt.subplots(1, 4, figsize=(12, 7))
# 表示するプロジェクションの名前を含むリスト
projections = ['最小プロジェクション', '平均プロジェクション', '最大プロジェクション', '標準偏差プロジェクション']
# 計算するプロジェクションの名前を含むリスト
np_projection = [np.min, np.mean, np.max, np.std]
# プロジェクションを計算する
for i in range(len(projections)):
h1_histone_3d = np.atleast_3d(np_projection[i](h1_histone, axis=0).astype(h1_histone.dtype))
pila_3d = np.atleast_3d(np_projection[i](pila, axis=0).astype(pila.dtype))
color_h1 = np.asarray([1, 0, 1]).reshape((1, 1, -1))
color_pila = np.asarray([0, 1, 0]).reshape((1, 1, -1))
h1_magenta = h1_histone_3d * color_h1
pila_green = pila_3d * color_pila
merged = np.clip(h1_magenta + pila_green,0,255)
axs[i].imshow(merged)
scalebar = ScaleBar(0.0721501, 'um', length_fraction=0.2, height_fraction=0.02, location='lower right')
axs[i].add_artist(scalebar)
axs[i].axis('off')
axs[i].set_title(projections[i])

5D画像(x、y、チャンネル、Zスライス、および時間)
共焦点5Dイメージングは、細胞および亜細胞レベルでのダイナミックなプロセスについての驚くべき洞察を提供する生物学の貴重なツールです。これらの画像には、x、y、zスライス、チャンネル、および時間が含まれており、時間的変化の視覚化と分析を可能にします。以下のコードでは、各時間点ごとにPilA-GFPとHistone H1-mRFP(マージされた画像)の最大投影を示すことで、共焦点5Dイメージングの重要性を示しています。各時間点は約20分を表しています。
# scikit-imageを使用して5Dイメージスタックを読み取る
img = sk.io.imread("3_channel_zstack_time_eisosome.tif")
print(f"データセットは{img.shape[2]} x {img.shape[3]}ピクセルで、{img.shape[4]}チャンネル、{img.shape[1]}スライス、{img.shape[0]}時間点を持っています")
# チャンネルを分離する
h1_histone = img[:,:,:,:,0]
pila = img[:,:,:,:,1]
brightfield = img[:,:,:,:,2]
# サブプロットを持つ図を作成する
fig, axs = plt.subplots(6, 7, figsize=(19,14))
# 投影を計算し(出力では同じdtypeを保持)、各時間点のマージドイメージを作成する
time = 0
for i in range(img.shape[0]):
# H1ヒストンチャンネルの最大投影を計算する
h1_histone_3d = np.atleast_3d(np.max(h1_histone[i,:,:,:], axis=0).astype(h1_histone.dtype))
# PilAチャンネルの最大投影を計算する
pila_3d = np.atleast_3d(np.max(pila[i,:,:,:], axis=0).astype(pila.dtype))
# 明るいフィールドチャンネルの最小投影を計算する
brightfield_3d = np.atleast_3d(np.min(brightfield[i,:,:,:], axis=0).astype(brightfield.dtype))
# 各チャンネルに対応する色を定義する
color_h1 = np.asarray([1, 0, 1]).reshape((1, 1, -1)) #マゼンタ色の1x1x3配列を作成する
color_pila = np.asarray([0, 1, 0]).reshape((1, 1, -1)) #緑色の1x1x3配列を作成する
color_brightfield = np.asarray([0.5, 0.5, 0.5]).reshape((1, 1, -1)) #グレーの1x1x3配列を作成する
# チャンネルを組み合わせてマージドイメージを作成する
h1_magenta = h1_histone_3d * color_h1
pila_green = pila_3d * color_pila
brightfield_gray = brightfield_3d * color_brightfield
merged = np.clip(h1_magenta + pila_green, 0, 255)
# 対応するサブプロットにマージドイメージをプロットする
axs[i // 7, i % 7].imshow(merged)
axs[i // 7, i % 7].axis('off')
axs[i // 7, i % 7].set_title(f"{time} 分")
scalebar = ScaleBar(0.0721501, 'um', length_fraction=0.2, height_fraction=0.02, location='lower right')
axs[5, 1].add_artist(scalebar)
time += 20
plt.tight_layout()
plt.show()
データセットは589 x 589ピクセルで、3チャンネル、4スライス、37時間点を持っています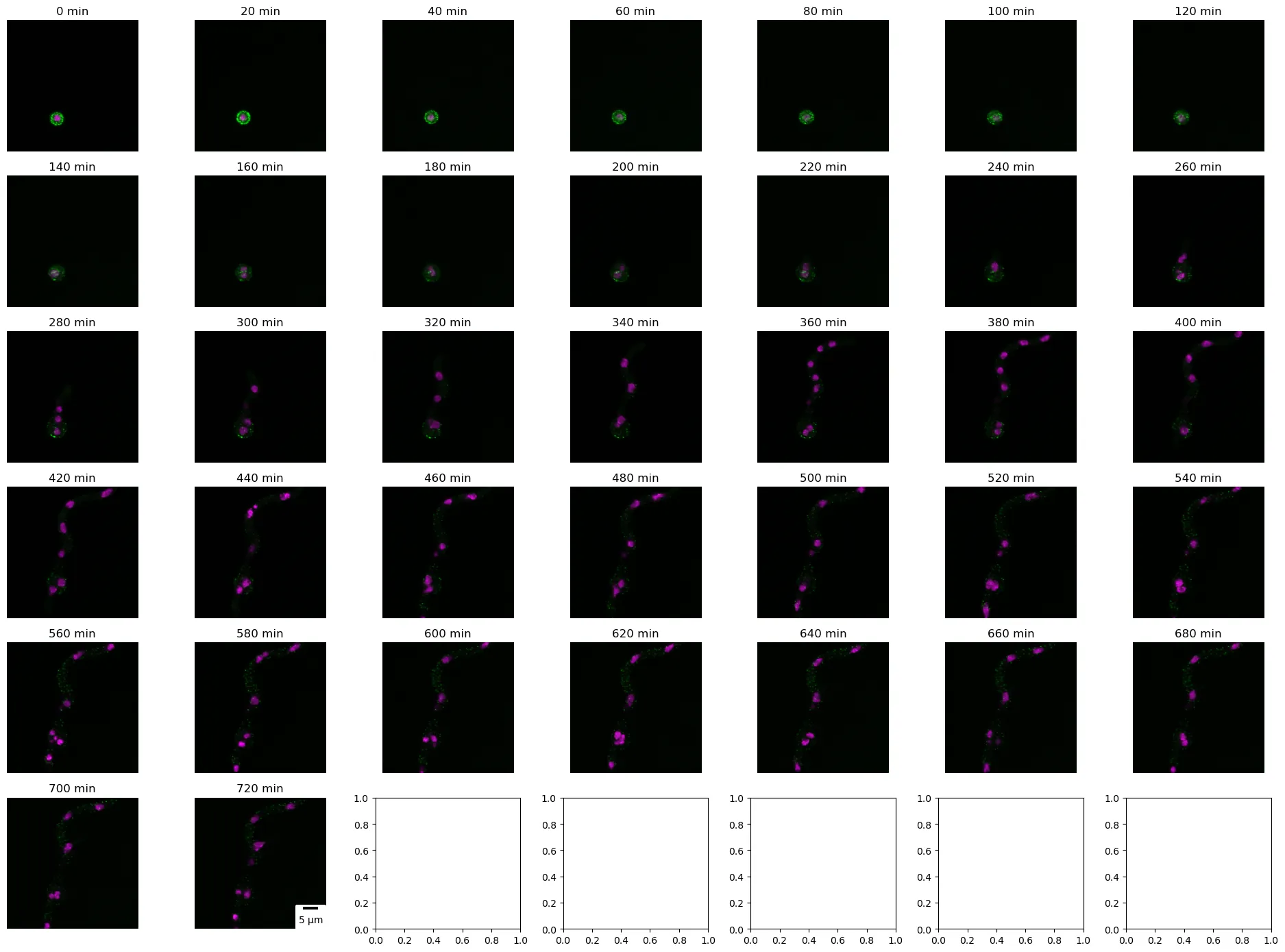
以下に、時間の経過に伴うA. nidulansの明るいフィールドスライスを表示しています。
# scikit-imageを使用して5Dイメージスタックを読み取る
img = sk.io.imread("3_channel_zstack_time_eisosome.tif")
# チャンネルを分離する
h1_histone = img[:,:,:,:,0]
pila = img[:,:,:,:,1]
brightfield = img[:,:,:,:,2]
# サブプロットを持つ図を作成する
fig, axs = plt.subplots(6, 7, figsize=(19,14))
# 各時間点の明るいフィールドチャンネルのスライスを選択する
time = 0
for i in range(img.shape[0]):
brightfield_2_slice = brightfield[i, 2, :, :]
# 対応するサブプロットに2つ目のスライスをプロットする
axs[i // 7, i % 7].imshow(brightfield_2_slice, cmap="gray")
axs[i // 7, i % 7].axis('off')
axs[i // 7, i % 7].set_title(f"{time} 分")
scalebar = ScaleBar(0.0721501, 'um', length_fraction=0.2, height_fraction=0.02, location='lower right')
axs[5, 1].add_artist(scalebar)
time += 20
plt.tight_layout()
plt.show()

結論
このチュートリアルでは、2D、3D、4D、および5Dを含むさまざまな次元の複雑な光学顕微鏡画像の読み取りと視覚化のプロセスについて詳しく説明します。matplotlibはデータの視覚化には強力なライブラリですが、マイクロスコープ画像などの3D、4D、および5Dの画像の視覚化には適していない場合があります。matplotlibは2D画像を効果的に処理できますが、より高次元の画像の視覚化は困難で制限があります。しかし、解決策はあります:
- Microfilm [9]、[10]は、マルチチャンネル画像のプロットを簡素化します
- そして、マイクロスコープ画像を含む多次元の科学データの視覚化と探索に特化した人気のあるPythonライブラリNapari [8]。Napariは、matplotlibの機能を超えるインタラクティブで多機能なインターフェースを提供します。
次回のチュートリアルでは、Napariについて詳しく説明し、高次元の画像の視覚化におけるその機能をより良く理解します。
このブログ投稿には、私のGitHubで閲覧できるJupyter Notebookを用意しました。

参考文献:
[1] C. T. Rueden and K. W. Eliceiri, “Visualization approaches for multidimensional biological image data,” BioTechniques, vol. 43, no. 1S, pp. S31–S36, Jul. 2007, doi: 10.2144/000112511.
[2] J. B. Pawley, “Points, Pixels, and Gray Levels: Digitizing Image Data,” in Handbook Of Biological Confocal Microscopy, J. B. Pawley, Ed., Boston, MA: Springer US, 2006, pp. 59–79. doi: 10.1007/978–0–387–45524–2_4.
[3] P. Bankhead, “Introduction to Bioimage Analysis — Introduction to Bioimage Analysis.” https://bioimagebook.github.io/index.html (accessed Jun. 29, 2023).
[4] J. Johnson, “Not seeing is not believing: improving the visibility of your fluorescence images,” Mol. Biol. Cell, vol. 23, no. 5, pp. 754–757, Mar. 2012, doi: 10.1091/mbc.e11–09–0824.
[5] I. Vangelatos, K. Roumelioti, C. Gournas, T. Suarez, C. Scazzocchio, and V. Sophianopoulou, “Eisosome Organization in the Filamentous AscomyceteAspergillus nidulans,” Eukaryot. Cell, vol. 9, no. 10, pp. 1441–1454, Oct. 2010, doi: 10.1128/EC.00087–10.
[6] A. Athanasopoulos, C. Gournas, S. Amillis, and V. Sophianopoulou, “Characterization of AnNce102 and its role in eisosome stability and sphingolipid biosynthesis,” Sci. Rep., vol. 5, no. 1, Dec. 2015, doi: 10.1038/srep15200.
[7] A. P. Mela と M. Momany、「Aspergillus nidulans の細胞内コンパートメント内でのヒストンH1の間核拡散」、PloS One、vol. 13、no. 8、p. e0201828、2018年、doi: 10.1371/journal.pone.0201828。
[8]「napari: Pythonにおける多次元画像の高速でインタラクティブなビューアー — napari」。https://napari.org/stable/(アクセス日:2023年7月4日)。
[9] G. Witz、「マイクロフィルム」。2023年6月27日。アクセス日:2023年7月14日。[オンライン]。利用可能:https://github.com/guiwitz/microfilm
[10]「Bio-image Analysis Notebooks — Bio-image Analysis Notebooks」。https://haesleinhuepf.github.io/BioImageAnalysisNotebooks/intro.html(アクセス日:2023年7月14日)。
We will continue to update VoAGI; if you have any questions or suggestions, please contact us!
Was this article helpful?
93 out of 132 found this helpful
Related articles
- ソースコード付きのトップ14のデータマイニングプロジェクト
- 「AIはデータガバナンスにどのように影響を与えているのか?」
- CMU、AI2、およびワシントン大学の研究グループが、NLPositionalityというAIフレームワークを導入しましたこれは、デザインのバイアスを特徴づけ、NLPのデータセットとモデルの位置性を定量化するためのものです
- 「Langchain x OpenAI x Streamlit — ラップソングジェネレーター🎙️」
- 「Google Sheetsにおける探索的データ分析」
- データサイエンティストのためのDockerチュートリアル
- 「データ分析のためのトップ10のAIツール」
