dtreevizを使用して、信じられないほどの意思決定木の視覚化を作成する
Create incredible decision tree visualizations using dtreeviz.
この便利なライブラリを使って決定木モデルを視覚化する方法
モデルの説明可能性に関して言えば、決定木は最も直感的かつ説明可能なモデルの1つです。すべての決定木モデルは、人間が理解できるルールのセットとして説明できます。決定木モデルを視覚化できることは、モデルの説明可能性のために重要であり、ステークホルダーやビジネスマネージャーがこれらのモデルに信頼を置くのを助けることができます。
幸いにも、我々はdtreevizライブラリを使用して簡単に決定木モデルを視覚化および解釈することができます。 この記事では、回帰および分類のツリーベースのモデルを視覚化するためにdtreevizを使用する方法を示します。
dtreevizのインストール
以下のコマンドを使用して、pipで簡単にdtreevizをインストールできます。
pip install dtreevizオペレーティングシステムによっては、インストールする必要がある依存関係や追加のライブラリの詳細なリストについては、このGitHubリポジトリを参照してください。
回帰木の視覚化
このセクションでは、糖尿病データセットで決定木回帰器をトレーニングします。このチュートリアルのすべてのコードは、このGitHubリポジトリで入手できます。このPythonコードを実行するための環境として、Jupyterを使用していることに注意してください。このチュートリアルで書いたすべてのコードは、このGithubリポジトリで見つけることができます。
ライブラリのインポート
以下のコードブロックでは、scikit-learn DecisionTreeモジュールとdtreevizを含むいくつかの一般的なライブラリを単純にインポートしました。
import numpy as npimport pandas as pdfrom sklearn.tree import DecisionTreeClassifier, DecisionTreeRegressorimport dtreevizデータの読み込み
糖尿病データセットはscikit-learnで利用可能なので、以下のコードを使用してデータセットをインポートし、特徴量とターゲット値をnumpy配列のXおよびyに格納することができます。
from sklearn.datasets import load_diabetesdiabetes_data = load_diabetes()X = pd.DataFrame(data = diabetes_data['data'], columns=diabetes_data['feature_names'])y = diabetes_data['target']決定木モデルのトレーニング
ツリーを視覚化しやすくするために、決定木の最大深度を制限し、以下のようにデータでトレーニングすることができます。
dtree_reg = DecisionTreeRegressor(max_depth=3)dtree_reg.fit(X, y)ツリーの視覚化
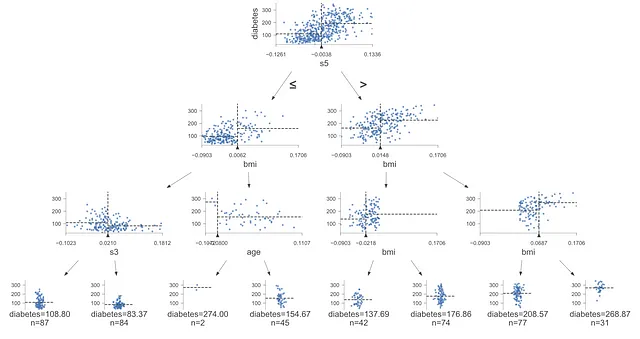
dtreevizの主要な機能の1つは、決定木モデルを視覚化できることです。以下のコードを使用することで、各ノードの決定境界を視覚的に表現したクールな決定木の視覚化を作成できます。
viz_model = dtreeviz.model(dtree_reg, X_train=X, y_train=y, feature_names=list(X.columns), target_name='diabetes')viz_model.view()
上記の視覚化では、各ノードの決定境界と特徴空間、各葉の回帰出力とサンプルサイズも表示されていることに注意してください。
葉の分布の視覚化
dtreevizが提供する別の便利な機能は、葉ノードの分布を視覚化する機能です。決定木の葉ノードには、各条件セットに応じて決定木が予測する実際の値が含まれます。 rtree_leaf_distributions関数を使用することで、回帰ツリーのためにこの視覚化を作成できます。
import matplotlib.pyplot as pltfrom mpl_toolkits.mplot3d import Axes3D%matplotlib inlineviz_model.rtree_leaf_distributions()
上記の可視化から、決定木は「糖尿病」というターゲット変数に対して268.9、208.6、176.9、137.7、154.7、274.0、83.4、または108.8を予測することができます。横に散らばった点は「糖尿病」というターゲット変数の値の分布を表し、小さな黒い線はその葉ノードでの予測に使用される平均値を表します。理想的には、葉の分布は分散が低いほどよく、予測に使用される平均値により多くの信頼性を持つことができます。
葉のサイズの可視化
以下の関数を使用して、葉のサイズ、つまり各葉ノードのサンプル数を可視化することもできます。
viz_model.leaf_sizes()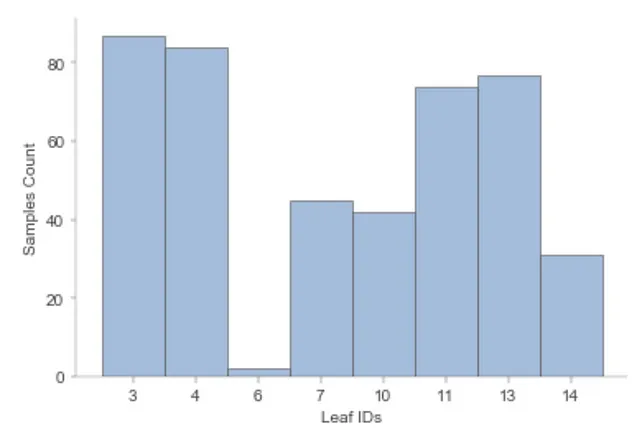
上記のプロットに基づくと、各葉のサンプル数を確認できます。この可視化は、回帰木の予測に自信を持つための良いツールです。
分類木の可視化
dtreevizを使用して分類木を可視化することもできます。この可視化は、回帰木の場合とはやや異なります。このセクションでは、Breast Cancer Wisconsinデータセットを使用して、決定モデルをトレーニングして可視化します。
データの読み込み
Breast Cancer Wisconsinデータセットはscikit-learnで利用可能なため、以下のコードを使用して読み込むことができます。
from sklearn.datasets import load_breast_cancercancer_data = load_breast_cancer()X = pd.DataFrame(data = cancer_data['data'], columns=cancer_data['feature_names'])y = cancer_data['target']決定木モデルのトレーニング
通常通り、scikit-learnを使用して決定木モデルをトレーニングすることは簡単です。最大ツリーの深さに制約を設けて、決定木を可視化しやすくすることもできます。
dtree_clf = DecisionTreeClassifier(max_depth=4)dtree_clf.fit(X, y)決定木の可視化
回帰木セクションと同じ関数を使用して、分類木を可視化することができます。ただし、可視化はやや異なります。
viz_model = dtreeviz.model(dtree_clf, X_train=X, y_train=y, feature_names=list(X.columns), target_name='cancer')viz_model.view()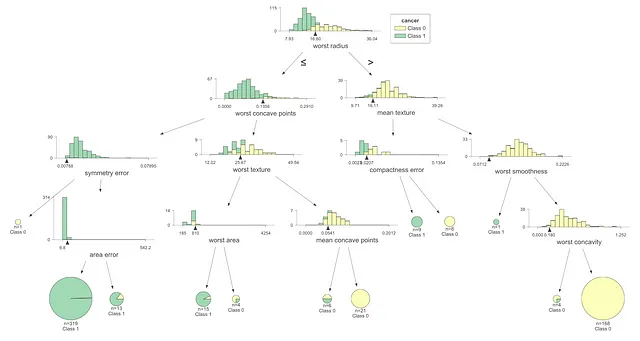
上記の分類木の可視化は、前のセクションでの回帰木の可視化とは異なります。選択された特徴量とターゲットが含まれる散布図は、ノードごとに色分けされたヒストグラムで表されます。
葉の分布の可視化
回帰木の葉分布の可視化と同じ関数を使用して、分類木の葉の分布を可視化することもできます。
viz_model.ctree_leaf_distributions()
各葉には、その葉のサンプルのクラスラベルの分布を示す積み上げ棒グラフが関連付けられています。ほとんどの葉は、1つのクラスに明らかに属するサンプルが多数含まれており、これは良い兆候であり、モデルの予測に自信を持つのに役立ちます。
特徴空間の可視化
下記の関数を使って、分類器の特徴空間を可視化することもできます。
viz_model.ctree_feature_space()
上記の特徴空間プロットは、分類木の学習精度、2つの特徴量の散布図、および2つのクラスを分離するために使用できる線形決定境界を示しています。
まとめ
ツリー型モデルの可視化に関しては、dtreevizは数多くの有用な可視化機能を提供する強力なライブラリです。この記事では提供される機能の一部しかカバーしておらず、dtreevizのGitHubリポジトリで読むことができる他の多くの機能があります。通常通り、この記事のすべてのコードは私のGitHubで入手できます。
メーリングリストに登録する
私のデータサイエンスコンテンツの更新情報を受け取るために、私のメーリングリストに登録してください。また、サインアップすると、ステップバイステップの機械学習問題解決ガイドが無料で提供されます!コンテンツの更新情報を受け取るために、Twitterでもフォローしてください。
そして、VoAGIコミュニティに参加して、数千人の他のライターからの記事を読んでみてください。
参考文献
- Terence Parr, dtreeviz: Decision Tree Visualization , (2023), GitHub.
We will continue to update VoAGI; if you have any questions or suggestions, please contact us!
Was this article helpful?
93 out of 132 found this helpful
Related articles
